【行业资讯】先导编辑开启基因治疗新纪元
自从2019 年首次发布这项基因编辑的新技术以来,先导编辑技术(prime editing,PE)发展迅速,并以其精准、高效的优势让人们期待越来越多的应用。Prime Medicine公司的Andrew Anzalone 和 David Liu 在2019年发表第一份报告,报告指出他们已经开发了下一代基因编辑系统,有利于使得先导编辑迈向基因编辑的最终目标:基因治疗。
先导编辑原理
由gRNA (pegRNA) 和与逆转录酶融合的Cas9 酶组成CRISPR/Cas9系统是先导编辑机制的重要组成部分。 当抑制Cas9酶两个核酸酶结构域之一,它就会变成“切口酶”,只能切割一条DNA链。因此,一旦gRNA 与目标的DNA序列结合,Cas9切口酶就会切割DNA的单链形成一个开口。
以gRNA的另一端作为模板;它的一部分与DNA的开口相辅相成,另一部分则进行所需的基因编辑。裂口与匹配位点结合后在逆转录酶添加下完成修复。编辑后的开口被重新整合到 DNA 中,并在互补链上的第二个切口的帮助下,在DNA 修复机制下修复匹配另一条链。
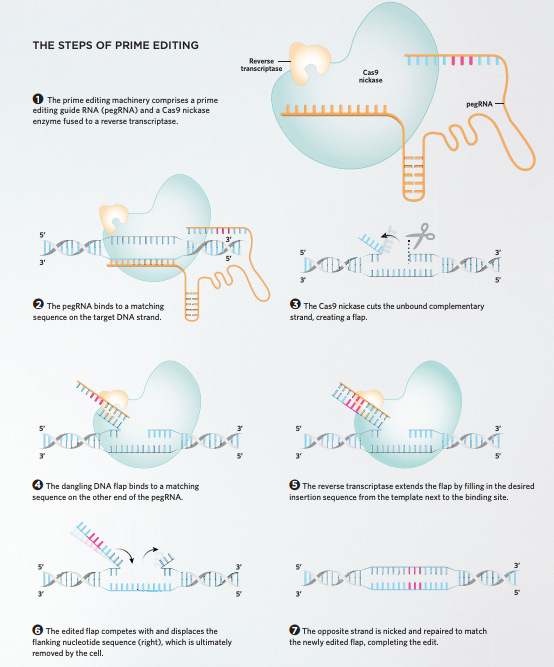
修改自© ISTOCK.COM, TTSZ; DESIGNED( ERIN LEMIEUX) 完整原图可联系下方客服
双先导编辑(twin prime): 下一代先导编辑器
最初的先导编辑器专注于比较小的点突变,再从单碱基替换到数十个碱基对长度的插入和删除的实现,但现在它无法修复更长的序列编辑或者是引起基因疾病的结构变异。针对这种情况Anzalone、Liu和他们的同事提出了双先导编辑。这个概念是以数学中的孪生素数猜想命名的,就像有无限多个素数一样,也有无限多个孪生素数,例如 17 和 19,它们是质数,仅相距两个。对于双先导编辑,Anzalone 和 Liu 使用了两种gRNA。通过创建两个在中间重叠的碱基序列,他们可以通过让一个 pegRNA 编辑前半部分、另一个编辑后半部分来编辑更大的DNA片段。
然而,双先导编辑仍然难以插入超过 100个碱基对的 DNA序列。为了打破这个限制,研究团队添加了一个额外的元件:位点特异性重组酶。在自然界中,这些酶识别并结合DNA中的特定位点,切割链并重新排列大片段,然后将链重新连接在一起。在基因组编辑中使用它们有利于优化双先导编辑的插入基因的能力。
于是他们决定使用双引物来添加酶的识别位点,然后整合一段供体DNA序列。 Prime Medicine公司将这种大型双先导编辑,用于提前辅助位点特异性整合酶进行基因编辑。 “它有可能实现基因大小的整合”Anzalone说。这套主要编辑器可以处理小型、中型和大型DNA序列的编辑。我们的下一个目标是找出如何使用这些工具来开发新的基因疗法。
先导编辑在基因疗法的应用
Anzalone认为:除了纠正点突变引起的相关遗传疾病之外,我们还在探索因为重复序列扩张引起的疾病,例如亨廷顿病和弗里德赖希共济失调,我们相信我们可以消除这些致病性重复序列并可能对患者群体产生影响,他还提到,先导编辑可用于生成有效的抗肿瘤CAR-T细胞。
Fyodor Urnov 认为:Prime Medicine 最紧迫的任务是尽快将他们的技术带给患者。他希望看到的不是Anzalone 和他的团队将技术开发到完美,而是将他们现有的技术转化为可以更快治愈患者的技术。在临床准备就绪之前可以优化技术的效力和安全性,但是这种想法对于临床医学领域来说并不正确。如果能满足监管标,并且对患者具有透明度就足够了。
Anzalone希望Prime Medicine 能够实现这两个目标并且设想未来能实现先导编辑的规模化应用,一种方法是“向染色体进军”,本质上是解决患者群体中更常见的一种基因突变,然后是更多的遗传疾病,这样就有希望获得合法的监管解决方案,而且这样的话,每个新的pegRNA 都不需要重新设计。 另一种方法是开发“长编辑序列”系统,该系统可以修复患者群体中可能发现的特定外显子中的所有突变,这样一种药品就可以治疗很多患者,而不仅仅是一个患者。
但是新技术的优先级和策略让人两难: 首先选择哪种疾病进行研究?谁先开始研究?这些问题很难简单回答。“对于早期的生物技术来说,最大的困难是弄清楚你应该做多少这样的事情,”Anzalone说。
★艾迪基因基于PE(Prime Editing)技术,全面升级研发Bingo™先导编辑点突变平台,在上千例基因编辑CRO项目经验中总结、优化、提升,成功率远超传统基因点突变方法。为广大科研企业提供精准、高效的基因点突变细胞构建服务。

Reference:
1. Kosicki M, et al. Repair of double-strand breaks induced by CRISPR-Cas9 leads to large deletions and complex rearrangements. Nat Biotechnol. 2018;36(8):765-771.
2.Anzalone AV, et al. Search-and-replace genome editing without double-strand breaks or donor DNA. Nature. 2019;576(7785):149-157.
3.Anzalone AV, et al. Programmable deletion, replacement, integration and inversion of large DNA sequences with twin prime editing. Nat Biotechnol. 2022;40(5):731-740.
4.Zhao Z, et al. Prime editing: advances and therapeutic applications. Trends Biotechnol. 2023.
18102225074(微信同号)
market@edgene.cn
最近资讯:



