【文献解读】CRISPR文库筛选解码人类原代T细胞的刺激反应

CRISPRa文库通过在细胞中激活特定基因,科学家们能够深入了解这些基因对细胞功能和生物学过程的调控作用。这有助于揭示基因与疾病之间的关联,为疾病的病因研究提供新的线索。小编精心挑选了一篇关于CRISPRa文库筛选的高分文章进行解读,看下去吧~
格莱斯顿—加州大学旧金山分校基因组免疫学研究所的科研团队在Science上发表题为CRISPR activation and interference screens decode stimulation responses in primary human T cells的文章,影响因子56.9。该研究报道了人类T细胞的全基因组CRISPR激活(CRISPRa)和干扰(CRISPRi)筛选,以识别控制白介素-2 (IL-2)和干扰素-γ (IFN-γ)产生的基因网络。研究人员将CRISPRa筛选与单细胞RNA测序相结合,揭示干扰调节T细胞激活和提升细胞状态的机制以及对关键免疫细胞功能进行重新编程的基因。

细胞因子在刺激反应中调节T细胞的活动,是平衡免疫反应的关键,细胞因子失调可能会导致癌症中的自身免疫、免疫缺陷和免疫逃避。白细胞介素-2 (IL-2)和干扰素-γ(IFN-γ)是调节T细胞的重要细胞因子,前者主要由CD4+T细胞分泌,使T细胞扩张,可应用在自身免疫和癌症治疗;后者可由CD4+和CD8+ T细胞分泌,与癌症免疫治疗相关。在本研究中,研究人员开发了人类T细胞CRISPRa和CRISPRi筛选平台,对控制人类T细胞的细胞因子产生途径和通路进行了研究。
传统的CRISPRa筛选使用的慢病毒传递速度较慢,仅限于小规模实验,要想大规模筛选干扰人类T细胞的基因和通路需要更高速的传递工具。为了突破这个局限性,研究人员开发了一种高滴度慢病毒,使用最小的dCas9-VP64载体(pZR112),两者结合使整体转导效率高达80%,有效增强CRISPRa-SAM系统对已建立的表面标记物靶基因诱导表达的强度。为验证该系统的实际效果,研究人员对超过18800个蛋白质编码基因和超过112000个sgRNAs进行全基因组CRISPRa筛选,使用流式细胞荧光分选技术(FACS)分离出能产生IL-2的CD4+ T细胞和产生IFN-γ的CD8+ T细胞,通过sgRNA定量证实,靶向IL-2和IFN-γ的sgRNAs在不同细胞因子中高度富集,非靶向对照的sgRNAs未明显富集,CRISPRa筛选在两种不同的人类献血者中具有高度的重复性,CRISPRa筛选IL-2和IFN-γ的基因水平统计分析分别显示444和471次命中,包括171次共享命中,表明该团队成功构建了一个可靠的大规模CRISPRa筛选平台。
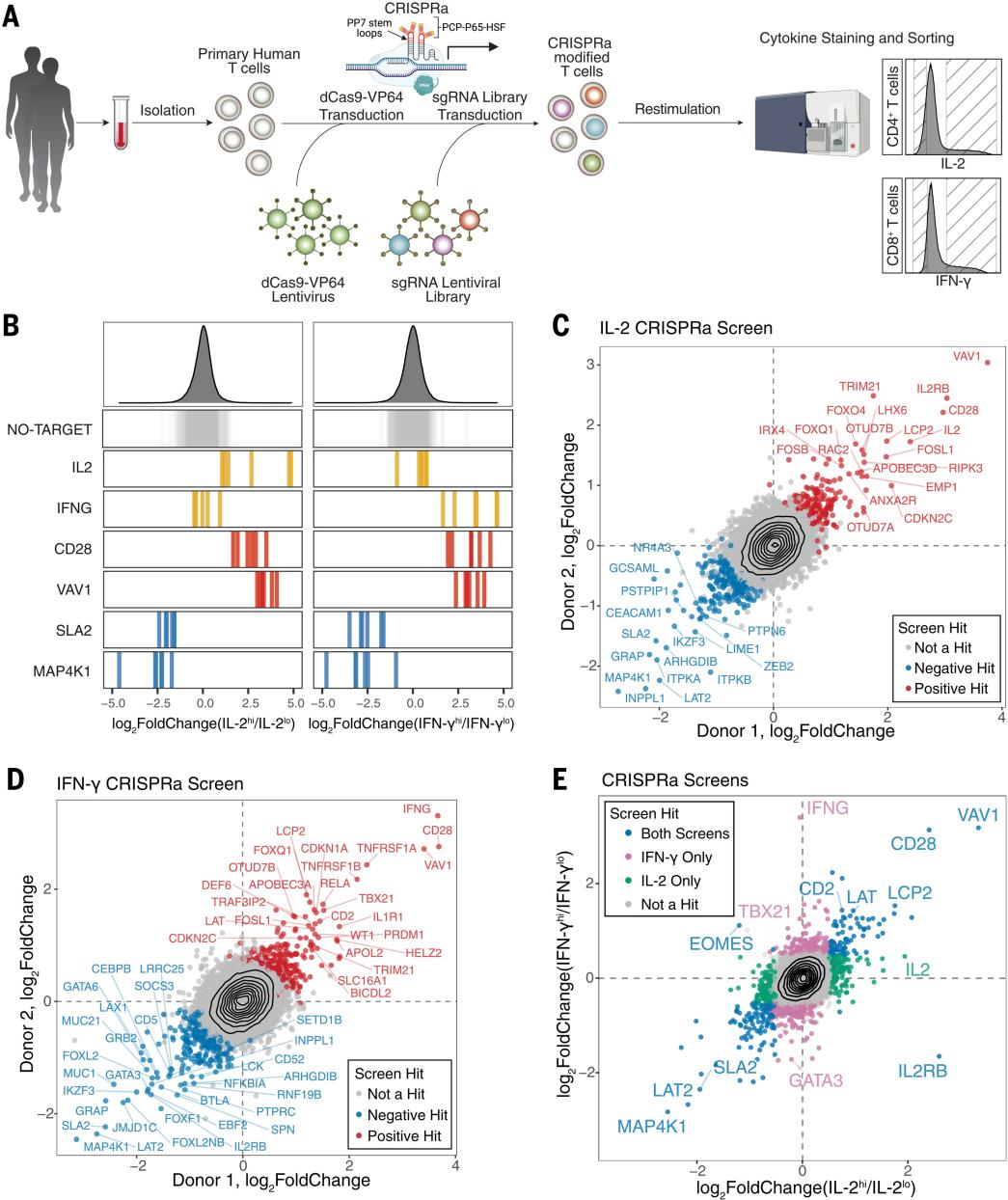
图1 全基因组CRISPRa筛选识别T细胞中IL-2和IFN-γ产生的调节因子
某些细胞因子只有使其功能缺失才能发现,为研究这些细胞因子,研究人员使用CRISPRi和优化后的慢病毒进行IL-2和IFN-γ筛选实验,识别出226和203个基因命中,包括92个共享命中。CRISPRi倾向于筛选高mRNA表达的基因,包括CD3复合体。而CRISPRi未能检测筛选到的低表达或完全不表达的调节因子,研究人员则使用CRISPRa进行识别筛选,成功筛选出某些T细胞环境下才诱导表达的调节因子,如PIK3AP1和IL1R1。此外,CRISPRi筛选了核因子кB(NF-кB) 通路的关键成分,该通路是产生IFN-γ的必需通路,T细胞刺激信号通过MALT1, BCL10, TRAF6和TAK1(由MAP3K7编码)抑制NF-кB复合体(该复合体由CHUK, IKBKB和IKBKG编码),促进IFN-γ的产生。CRISPRa则显示一组阳性的IFN-γ调节因子,包括TNFRSF和IL1R1,这些调控因子也经过NF-кB通路但没有被CRISPRi检测到,因此CRISPRa和CRISPRi互补可以全面发现功能性细胞因子调控因子。
研究人员通过KEGG进行基因富集分析,深入了解通过CRISPRi和CRISPRa筛选富集的功能通路。分析大量全基因组关联研究的数据,发现IFN-γ的CRISPRi和CRISPRa调节因子以及IL-2的CRISPRa调节因子都位于免疫遗传性状丰富的区域;整合分析CRISPRa和CRISPRi筛选出来的细胞因子,其中一些基因在两种筛选中都被成功识别,说明这些基因是T细胞生成的刺激反应性细胞因子中的核心调节因子;包括PTPRC (CD45)在内的少数靶基因,CRISRPa和CRISPRi都以相同的方向影响细胞因子的产生,表明激活和干扰共同影响某些基因的最佳表达水平。研究人员利用这些综合数据结合文献综述构建了细胞因子产生的调节信号通路图,包括钙信号通路基因和细胞因子信号通路基因,同时研究人员使用CRISPRa还识别出了在以往的文献中缺失的调控因子,表明CRISPRa和CRISPRi互相补充进行筛选能更精确全面的绘制细胞因子产生的调节信号通路。

图2 互补CRISPRa和CRISPRi筛选全面揭示了T细胞中细胞因子的产生通路
为了在筛选命中中获得更深入的表型表征,研究人员选择14个不同种类的筛选命中进行了CRISPRa阵列实验。研究人员通过CD4+和CD8+ T细胞的细胞内染色来评估IL-2、IFN-γ和TNF-α,结果显示14个靶基因中有13个基因导致相关细胞因子的阳性细胞比例发生显著变化,全阵列sgRNA组细胞内细胞因子染色除了TNFRSF1A、IL2和IFN-γ,其他调节因子在没有刺激的情况下不会自发引起细胞因子的产生。在T细胞分化过程中,FOXQ1和TNFRSF1A显著降低了CD62L+细胞的百分比,表明细胞因子向效应T细胞的转移是一种潜在的机制。研究人员测量了48种分泌的细胞因子和趋化因子,在确认细胞因子对IL-2、IFN-γ和TNF-α的影响与细胞内染色基本一致后,对所有细胞因子进行了主要成分分析和分层聚类,其中VAV1和FOXQ1导致了1型特征细胞因子的增加和2型细胞因子的抑制;OTUD7B作为近端TCR信号的正向调节因子明显增加了2型细胞因子。研究人员对FOXQ1和对照组sgRNA CD4+ T细胞进行了批量RNA测序(RNA-seq),发现其与分泌体效应有很强的相关性,识别出的调节因子不仅能调节TCR刺激和信号转导,还能调节T细胞分泌体。
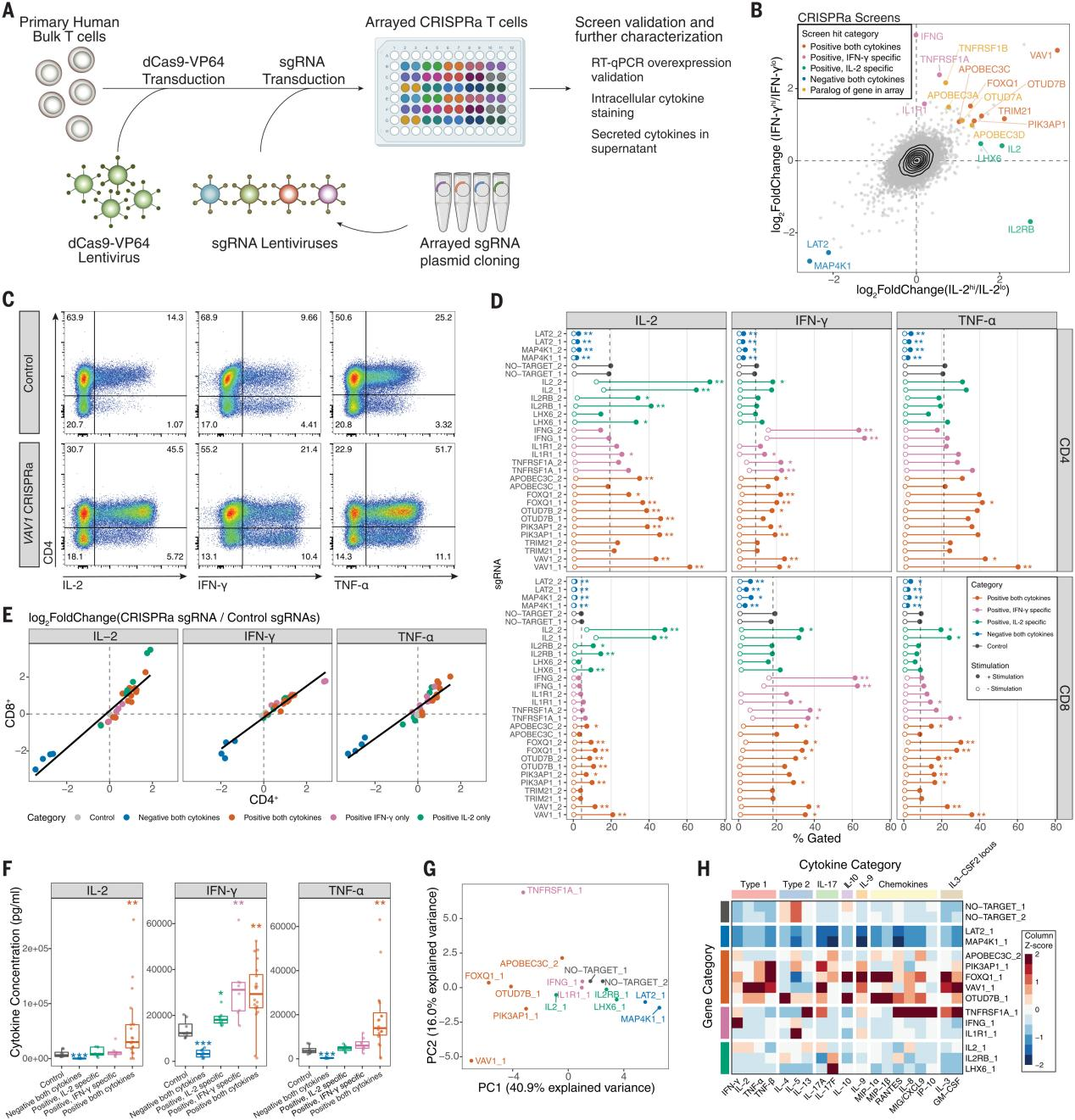
图3 通过阵列分析CRISPRa筛选命中的表征
研究人员开发了CRISPRa Perturb-seq平台用于评估每个CRISPRa基因诱导产生的整体分子签名,对大约56000个原始人类T细胞的刺激反应调控因子进行分析,证实sgRNAs可导致其靶基因表达显著增加。研究人员检测CRISPRa扰动的激活分数,除了IKZF3(编码Aiolos转录因子)外,其余负调控因子均降低了激活分数,表明它们可以广泛减弱刺激的强度,而IKZF3降低了IFN-γ的表达,但并未降低整体激活评分,提示可能存在一种不同的细胞因子基因调控机制。
研究人员使用簇分析来描述CRISPRa-driven细胞在二次刺激和静息T细胞中的细胞状态,鉴定了上调程度最高的基因表达标记物和细胞因子基因,揭示了CRISPRa促进的T细胞状态的多样性。研究人员还发现了两个表达IFN-γ的不同的细胞簇(簇1和12),簇1被标记为CCL3和CCL4高表达,并被sgRNAs富集;簇12富集了激活NF-кB通路的sgRNAs。这些结果表明增强刺激/共刺激可能驱动T细胞进入激活的IFN-γ表达状态,一个TNFRSF受体基因亚群的激活也促进了细胞状态。因此,CRISPRa Perturb-seq揭示了细胞因子产生的调节因子是如何调节T细胞激活并使细胞进入不同的刺激响应状态。
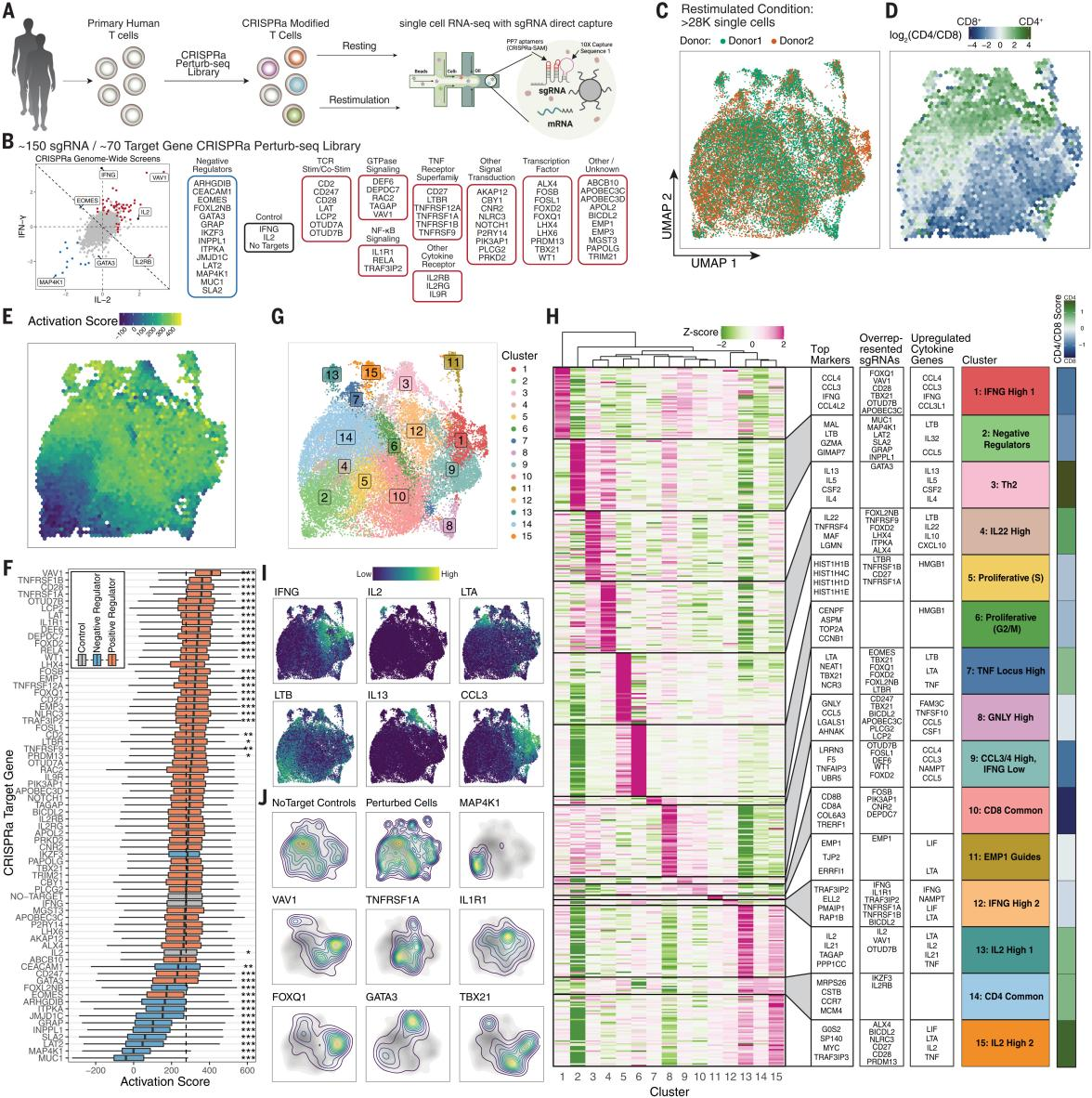
图4 CRISPRa Perturb-seq捕捉由全基因组细胞因子筛选所驱动的不同T细胞状态
综上所述,研究人员通过CRISPRi确定了所需的细胞因子调节因子,通过CRISPRa揭示了通路功能中的关键信号瓶颈,以及在体外培养的T细胞中不一定活跃的调节因子。本研究中开发的技术将使筛选人类初级T细胞和潜在的其他初级细胞类型的方法成为可能,T细胞CRISPRa和CRISPRi筛选平台将有助于对控制人类T细胞的细胞因子产生途径和通路的研究。
艾迪基因提供CRISPR文库筛选一站式完整解决方案,文库设计、定制、包慢病毒、正负筛选等一系列服务均可提供。还有90+种文库质粒/病毒等你来选,下单即筛,加速您的科研进程!
近期资讯
【前沿资讯】Prime Editing新动向-点突变细胞助力基因治疗临床前研究
联系我们
18102225074(微信同号)
market@edgene.cn



