【文献解读】基于CRISPR/cas13a的等温一锅式RNA检测平台---SATCAS

快速、经济、灵敏的病原体核酸检测在临床诊断和感染管理中起着至关重要的作用。传统的基于CRISPR/Cas13a的检测方法在分子诊断中显示出巨大潜力,但是大多数需要一个转录步骤,并且多步骤的开放式操作容易受到污染,限制了它们的广泛应用。为了克服现有技术的局限性,北京大学深圳医院与北京大学人民医院联合复旦大学的研究人员提出了一种等温一锅式RNA检测平台---SATCAS,为临床诊断提供了更有效的病原体和SNPs检测。相关研究成果“SATCAS: A CRISPR/Cas13a-based simultaneous amplification and testing platform for one-pot RNA detection and SNPs distinguish in clinical diagnosis”近期发表在Biosensors and Bioelectronics(IF:10.7)杂志上。

血清乙型肝炎病毒 (HBV)作为一种高威胁力的传染性病原体急需快速有效的诊断方法进行抑制,其中针对病原体RNA的检测有着重要的意义。血清乙型肝炎病毒 (HBV) 的前基因组 RNA (pgRNA) 可作为反映 HBV 转录活性的替代标记物 。然而,目前很少有用于量化乙型肝炎疾病患者血清HBV RNA的标准化检测方法。基于CRISPR的分子诊断显示出良好的检测性能和低设备成本等优势,具有POCT的潜力,但是很少有CRISPR-Dx能够快速和特异性地检测活细菌病原体。对此,该研究将 simultaneous amplification and testing(SAT)与 CRISPR/Cas13a 相结合构建了一锅等温核酸检测平台 SATCAS,其能够在 40 分钟内直接快速检测每个反应中接近单拷贝的 RNA 分子。
一、SATCAS平台的工作原理
SATCAS(Simultaneous Amplification and Testing platform based on Cas13a)平台通过级联酶促反应过程起作用,它由两条引物、逆转录酶、T7 RNA聚合酶、CRISPR-Cas13a 及crRNA系统组成。当存在靶标RNA时,T7启动子引物与目标RNA结合,通过逆转录酶的作用生成RNA/DNA杂交双链。逆转录酶的RNase H活性特异性消化杂交的RNA链,留下含有T7启动子的DNA单链。接着第二条引物与DNA单链结合,并通过逆转录作用生成含有T7启动子的双链DNA。之后,T7 RNA聚合酶识别双链DNA中的T7启动子序列,并从该模板连续转录出RNA。最终,转录出的RNA被Cas13a识别,激活其切割活性,切割荧光报告分子,发出可检测的荧光信号。
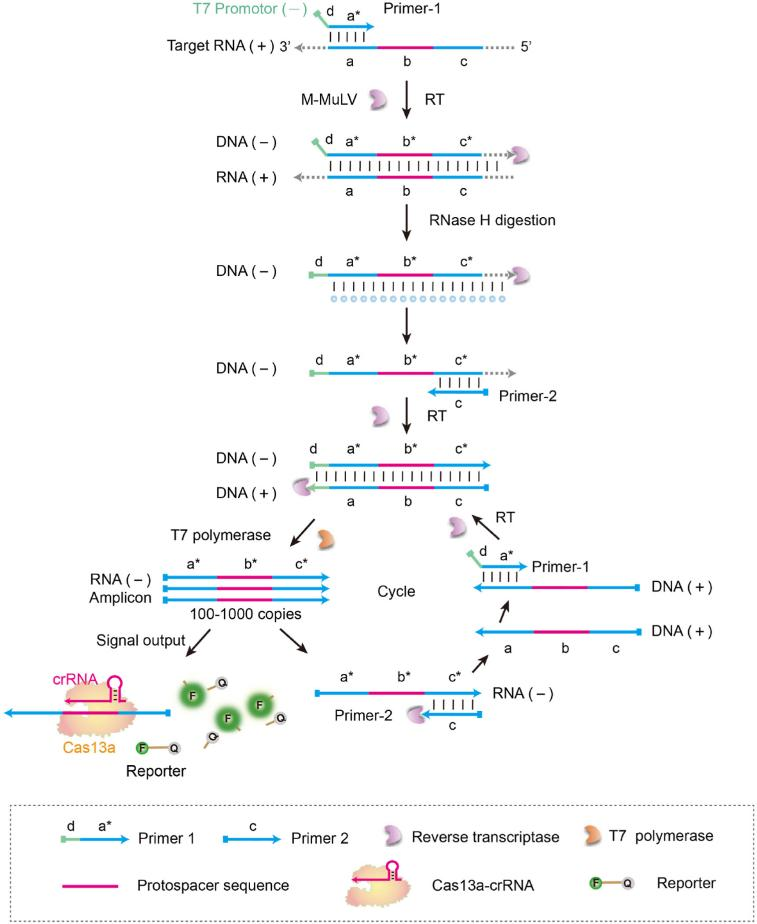
图1 SATCAS检测原理
二、一锅式检测的探索与SATCAS可行性验证
为了避免两步操作过程中的污染,研究人员尝试将SAT反应与Cas13a介导的切割反应系统集成,但效果欠佳。经过多次优化后,他们最终决定将Cas13a系统与SAT扩增系统分开,分别置于试管底部和盖子中,并确定其检测流程为:在SAT扩增30分钟后,通过振荡将Cas13a切割系统与SAT产物混合,然后在37°C下继续进行5-10分钟的Cas13a切割反应,最后通过肉眼在蓝光下观察荧光结果。
此外,为了评估SATCAS的可行性,研究人员以SARS-CoV-2片段作为DNA模板进行了琼脂糖凝胶电泳和实时荧光测量实验,结果显示只有靶标存在下的完整CRISPR/Cas13a体系出显著的荧光信号,证明SATCAS检测的可行性。通过使用HBV特异性的SATCAS检测方法来检测其他常见病毒RNA,结果发现SATCAS对干扰病毒RNA几乎没有荧光反应,这证明了SATCAS具有高特异性。
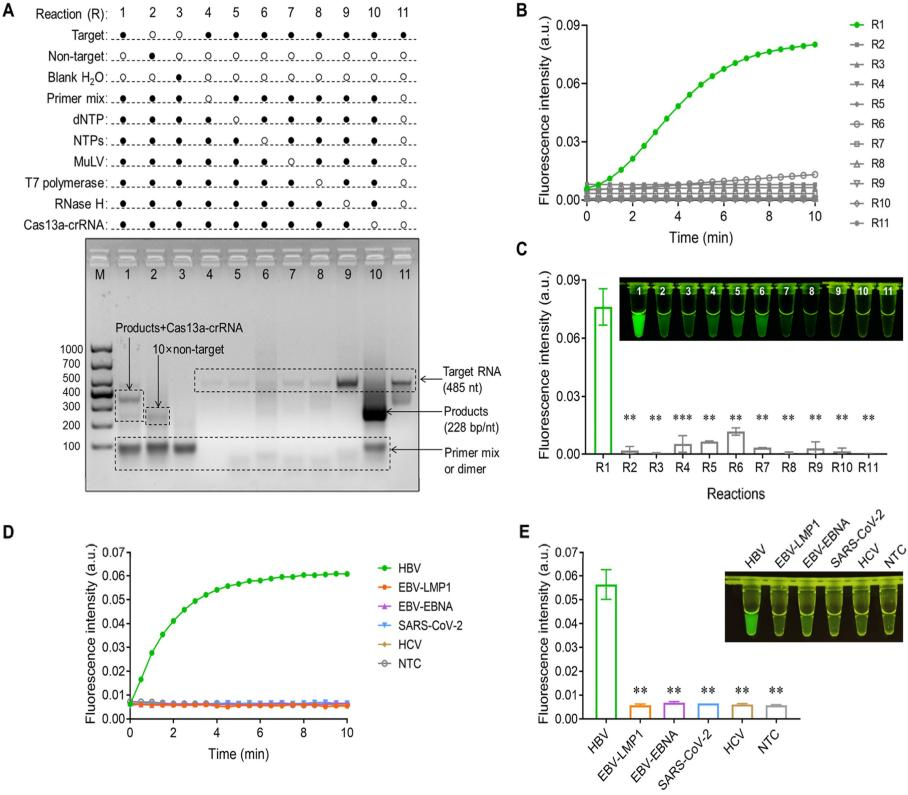
图2 SATCAS的可行性验证
三、活性细菌检测
RNA 检测是检测活体病原体的唯一方法。为了验证 SATCAS 区分活体和死亡细菌的能力,研究人员准备了不同比例的活体与死亡大肠杆菌(ATCC25922 E. coli)悬液样本进行检测,活体与总细菌的比例从 0% 到 100% 不等。他们首先使用标准平板计数法和 qRT-PCR 对样本进行分析,结果发现没有显著差异,这归因于所检测的靶标 DNA 在活体和死亡细菌中都存在。随后,他们使用 SATCAS 方法对这些样本提取的总RNA 进行分析,发现随着活体细菌比例的增加,荧光强度增加,表明 SATCAS 方法能够定量确定活体细菌的数量。
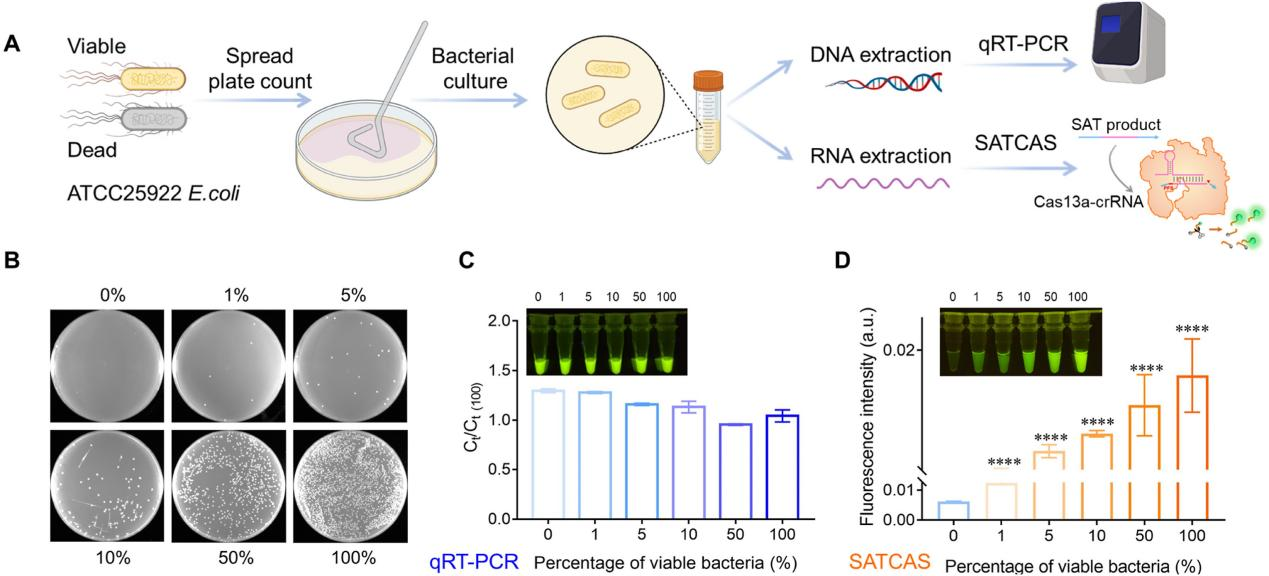
图3 活致病菌的定量
四、crRNA筛选和灵敏度试验
为了获得更好的SATCAS检测灵敏度,研究人员对crRNA进行优化筛选。他们设计了靶向 SARS-CoV-2 扩增片段的crRNA 序列,并分别测试所对应反应的实时荧光值和观察荧光强度可视化结果,结果确定crRNA5效果最好。依据筛选出的最优 crRNA5,研究人员对不同浓度梯度的 SARS-CoV-2 RNA 进行了检测,以确定 SATCAS 方法的灵敏度。测试结果显示,SATCAS 能够在 40μL 反应体系中一致检测到1×10−10 nM (0.1 aM) 的目标RNA,表明其检测限(LOD)为 2.41 RNA 拷贝/40 μL,与 qRT-PCR 和 LAMP 相当,且优于一些先前报道的基于 CRISPR 的方法。类似地,研究人员也对 HBV(乙型肝炎病毒)的 crRNA 进行了筛选,确定了最优的 crRNA2和其检测灵敏度。
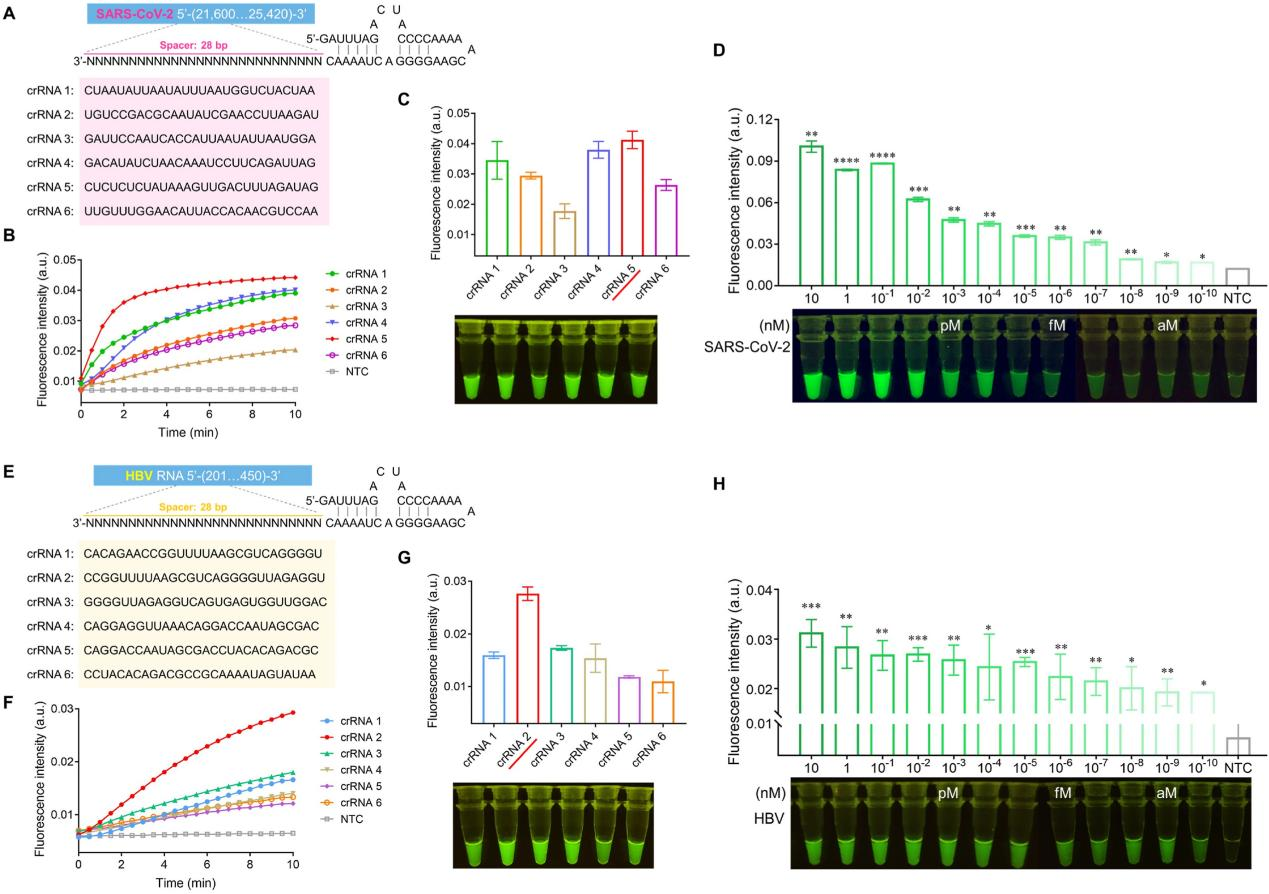
图4 crRNA的优化和SARS-CoV-2与HBV检测灵敏度的评估
五、SATCAS的特异性评估
LwaCas13a 在其识别区域的第 14 个位置表现出高达 70% 的单核苷酸突变鉴别能力,但通常对单核苷酸错配具有容忍性,这在区分 SNPs 时构成了挑战。为了评估 SATCAS 针对 HBV 药物抗性突变位点 L180M 的特异性,研究人员设计了一系列 crRNA,通过在 crRNA 的识别区域第 14 个位置引入 L180M 突变位点,并测试了这些 crRNA 对野生型(WT)和 L180M 突变目标的检测效果。终点荧光值和荧光可视化强度的比较结果显示,某些 crRNA 针对 WT 和突变目标显示出显著不同的检测强度,表明它们具有较高的特异性比率。进一步验证 SATCAS 在检测 HBV 药物抗性相关的酪氨酸-甲硫氨酸-天冬氨酸天冬氨酸(YMDD)变异体方面的单核苷酸突变鉴别能力,研究人员发现这些变异体可能导致病毒对拉米夫定产生抗性,导致病毒重新出现、复制甚至疾病进展。
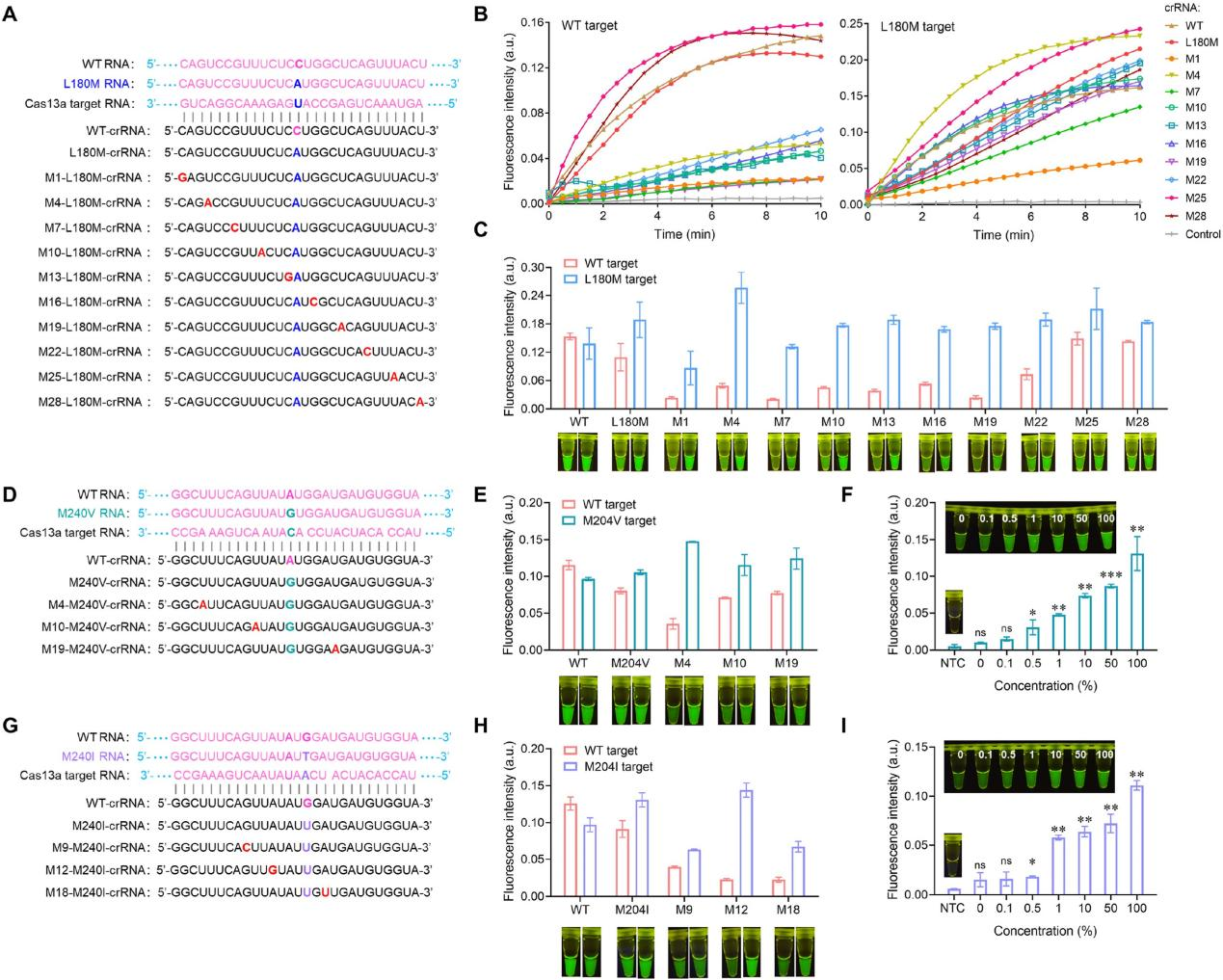
图5 SATCAS的特异性研究及其在HBV耐药YMDD突变鉴定中的应用
六、应用能力评估
为了评估SATCAS 方法在检测药物抗性突变方面的应用能力,研究人员分别对HBV YMDD 变异体、不同crRNA突变模板和不同浓度突变模板进行检测验证,所有结果都展示了SATCAS 方法能够准确、灵敏地检测药物抗性突变。此外,研究人员也评估了SATCAS 方法检测HBV 和 EBV 病毒的应用能力,他们收集了 HBV 感染患者的血液样本,并使用 SATCAS 方法和 qRT-PCR 方法进行了平行检测,对比结果发现两种方法的结果具有很高的一致性,SATCAS 显示出 100% 的灵敏度,92.86% 的特异性和 97.06% 的准确性。对其进行相关性分析,发现两者之间有强相关性或中等程度的相关性。通过与临床样本的比较,结果进一步支持了 SATCAS 方法在病毒感染诊断中的高灵敏度、特异性和稳健性,并验证了其在 RNA 检测中的应用潜力。
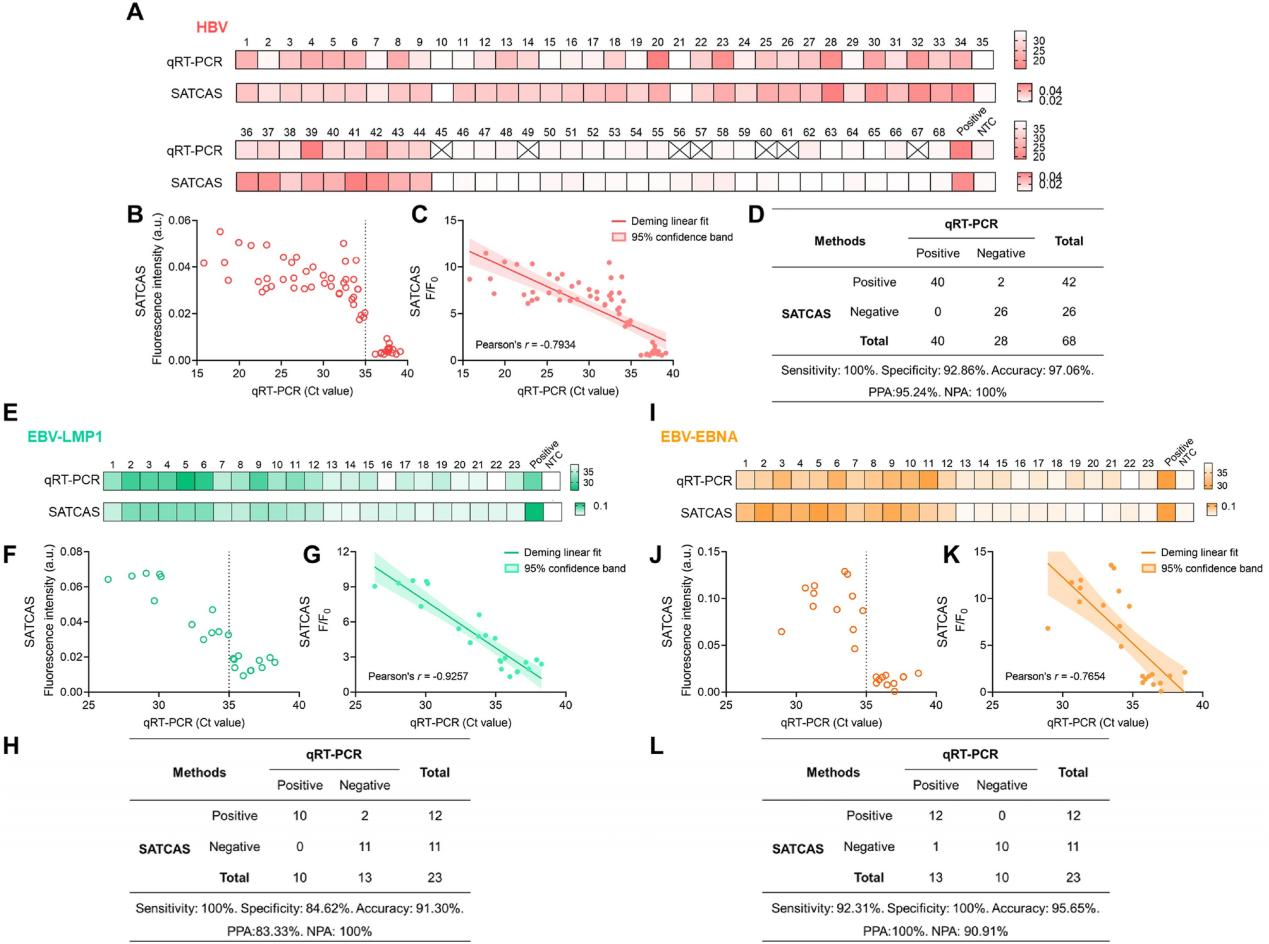
图6 SATCAS实际应用能力评估
综上,研究人员基于检测和区分与药物抗性相关的病毒突变的需求,将SAT与 CRISPR/Cas13a 相结合开发了一个等温一锅式RNA检测平台---SATCA,并为我们展示了该平台具有超灵敏、强抗干扰、操作简便、可视化的核酸检测能力,适用于活体病原体检测和病毒感染的临床诊断。尽管 SATCAS 仍需要从临床样本中提取 RNA 的额外步骤,但其展示了作为常规定量 RNA 检测方法的潜力。
原文链接:https://doi.org/10.1016/j.bios.2024.116636
艾迪基因深耕CRISPR/Cas基因编辑技术,基于蛋白质纯化平台,创新一步法提纯Cas酶,实现纯度、活性与回收率显著提升,性能远超国际同类产品,灵敏度达amol级,广泛应用于CRISPR研发、基因与疫病检测、食品安全检测等领域。更有恒温快速扩增试剂盒、CRISPR检测试剂盒现货,设备要求低,反应时间短,下单即达!
近期资讯
1.【文献解读】CRISPR KO筛选揭示了化疗耐药性的关键基因
2.【前沿资讯】CRISPR技术新动态--CRISPR/Cas9基因敲除细胞技术助力开发消融癌细胞新方法
3.【文献解读】CRISPR-Cas12a表现出金属依赖的特异性转换

联系我们
18102225074(微信同号)
market@edgene.cn



